판독 불가능했던 ‘의미 불분명 변이’ 42% 재분류
희귀질환· 유전성 암에 맞춤 치료 가능성 제시

국내 연구진이 인공지능(AI)을 활용해 유전자 검사에서 판독이 까다로웠던 ‘종결 코돈 변이’의 병원성을 정밀하게 예측하는 데 성공했다. 그간 질병과의 연관성을 규명하지 못해 ‘의미 불분명 변이’로 남겨졌던 영역을 AI로 정교하게 해석해낸 것이다.
연세대 강남세브란스병원 진단검사의학과 윤지훈·이경아 교수는 종결 코돈 변이의 병원성을 높은 정확도로 예측하는 AI 기반 유전자 변이 판독모델 TAILVAR를 개발하여 5일 밝혔다.
종결 코돈(Stop codon)은 세 가지 염기의 조합(TGA, TAG, TAA)으로, 우리 몸속 세포의 단백질 합성을 멈추게 하는 일종의 ‘정지 신호’다. 정상적인 경우 이 신호에 맞춰 단백질이 일정한 길이로 만들어지지만, 유전자 서열에 변이가 생겨 이 신호가 사라지면 단백질 말단(C-terminal)이 비정상적으로 길어진다.
이렇게 변형된 단백질은 길어진 서열로 인해 세포 내에서 엉겨 붙어 독성을 유발하거나, 세포 보호를 위한 비정상 단백질 분해 시스템에 의해 제거됨으로써 본래의 기능을 잃게 된다. 이는 결과적으로 도파민 반응 이상운동증, 뮤코다당증과 같은 희귀 질환은 물론, 유방암과 대장암 등 다양한 유전성 암을 유발하는 원인이 된다.
문제는 이러한 변이 중에는 실제 병을 일으키는 병원성 변이와 인체에 영향을 미치지 않는 양성 변이가 섞여 있는데, 이를 정확히 판독하기가 매우 어려웠다는 점이다. 이 때문에 많은 변이가 ‘의미 불분명 변이(VUS)’로 분류되어 환자 진단 및 치료에 활용되지 못했다.
이에 연구팀은 단백질 말단이 연장되는 종결 코돈 변이의 생화학적 특성에 주목했다. 단백질 말단부의 길이, 아미노산 구성, 소수성, 응집성 등 질병 발생과 밀접한 37가지 정밀 지표를 머신러닝에 학습시켜 TAILVAR 모델을 구축했다.
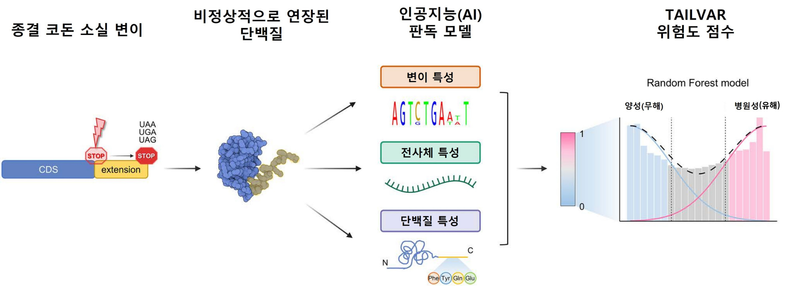
모델의 개발 및 성능 검증을 위해 백만 명 이상의 대규모 공개 유전체 데이터 베이스를 활용했으며, 그 결과 TAILVAR는 예측 정확도(AUROC) 0.956이라는 수치를 기록하며 현존하는 예측 도구 중 최고 수준의 성능을 입증했다. 실제 세포 실험 데이터와의 상관관계 분석에서도 가장 높은 상관성(ρ=0.379)을 나타내며, AI의 예측이 실제 생물학적 현상과 잘 맞아떨어짐을 증명했다.
특히 판독 불가능 상태였던 의미 불분명 변이(VUS)에 적용한 결과, 약 42%의 변이를 병원성 등으로 명확히 재분류하는 성과를 거뒀다. 이는 AI 모델이 단순히 수치를 계산하는 수준을 넘어, 실제 암 억제 유전자(APC, MLH1, SMAD4, BAP1 등)의 기능적 소실을 실험 데이터와 일치하게 예측해낼 수 있음을 시사한다.
윤지훈 교수는 “이번 연구는 그동안 평가하기 어려웠던 종결 코돈 변이를 체계적으로 분석할 수 있는 새로운 기준을 제시한 것에 의의가 있다”며 “아직 밝혀지지 않은 질병 원인 유전자와 변이 발굴을 가속화해, 정밀 의료와 개인 맞춤형 치료 전략에도 도움이 될 것이라 기대한다”고 밝혔다.
연구팀은 해당 데이터베이스를 오픈 소스 플랫폼 (https://github.com/dr-yoon/TAILVAR)을 통해 공개해, 모든 연구자들이 유전자 변이 해석 및 임상 판독에 활용하도록 했다.
이 연구는 생명과학 분야 국제 학술지 ‘Nucleic Acids Research’(IF 13.1)에 게재됐다.
김준영 기자 kjy1230@medifonews.com
< 저작권자 © Medifonews , 무단전재 및 재배포금지 >
- 본 기사내용의 모든 저작권은 메디포뉴스에 있습니다.
